| 时间:2023-03-17 |
2月15日,Caleb A. Lareau团队在nature protocols上发表了关于线粒体单细胞ATAC-seq的新方法,论文题目为 Mitochondrial single-cell ATAC-seq for high-throughput multi-omic detection of mitochondrial genotypes and chromatin accessibility。该方法可同时实现高通量线粒体DNA(mtDNA)基因分型和可及性染色质的分析。
在单细胞多组学方面,对整个体细胞的突变进行全基因组测序是相对昂贵的,并具有一定的挑战性,通常不与细胞状态的高维表征配对。Caleb A. Lareau团队开发了一种单细胞多组学方法“线粒体DNA单细胞转座酶染色质可及性测序技术(mtscATAC-seq)”(https://www.nature.com/articles/s41596-022-00795-3#Abs1)。以映射人体组织中细胞之间的克隆关系,研究线粒体遗传学的基本方面。
在与体细胞核基因突变相比较,mtDNA突变的检测具有一定的优势。第一,线粒体基因组很小(16.6kb),但足以提供实质性的遗传多样性。第二,它的突变率高于核DNA(高达10-100×)[1][2]。第三,线粒体基因组每个细胞的拷贝数很高(根据细胞类型,有数百到数千个),mtDNA的突变通常达到高水平的异质或同质。这种多拷贝数极大地促进了单细胞水平上更高异质的变体的检测。第四,同时检测细胞状态信息(例如,转录组和/或可及性的染色质分析)很容易与mtDNA基因分型兼容,为实现细胞(亚)克隆的细胞分型和基因组表征提供了宝贵的额外信息。
传统的scATAC-seq专注于分离没有线粒体的核基因,为了将线粒体和mtDNA保留在宿主细胞中,Caleb A. Lareau团队将固定交联与较温和的裂解条件相结合,来实现Tn5转座酶的核内和线粒体联合作用[3]。在同时标记核可及性的染色质和mtDNA后,通过10x Chromium Next GEM单细胞ATAC平台实现单细胞区划分和库生成(图1)。
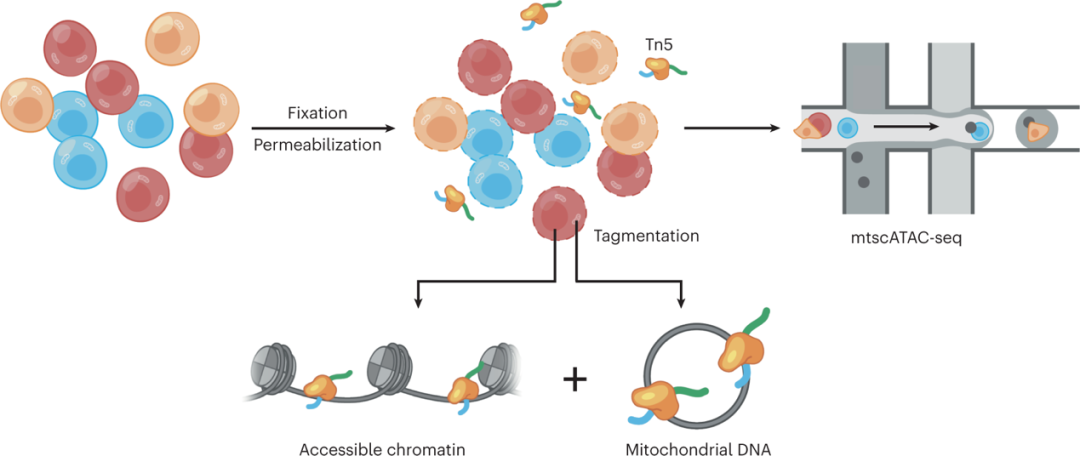
图1.mtscATAC-seq原理
为了促进研究影响线粒体和细胞代谢和功能的病原性mtDNA突变基因组的适应性变化结果,提出了计算mtscATAC-seq分析的示意图(图2)。Caleb A. Lareau团队还开发了线粒体基因组分析工具包(mgatk),可以对mtscATAC-seq数据快速进行生物信息处理,以识别体细胞mtDNA突变,可以识别到频率低至人类种群的0.005-0.01%的突变。
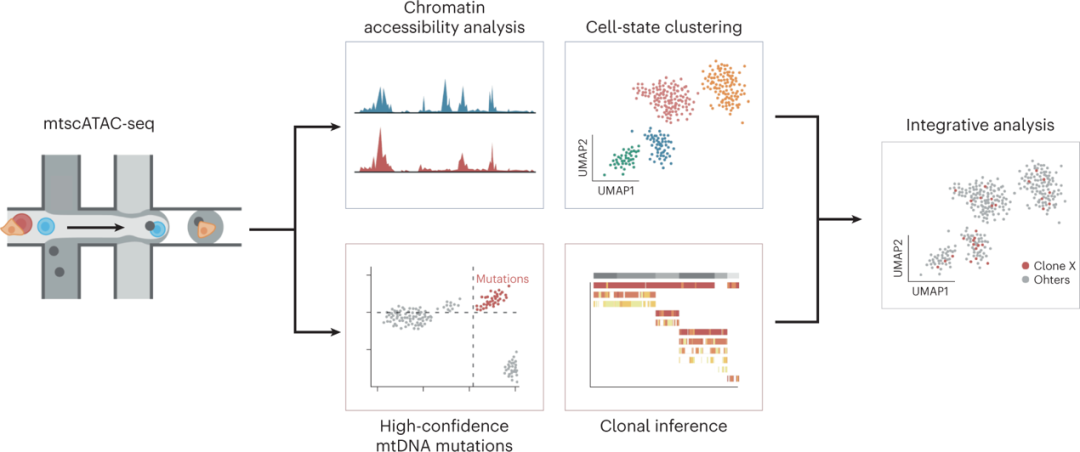
图2.mtscATAC-seq分析流程示意图
mtscATAC-seq可以从来自复杂细胞或组织的单个细胞中分析全基因组染色质的可及性,并且可以以扩展的方式同时与线粒体基因型一起分析,也能够通过增强或修改mtscATAC-seq实验流程来探索其它实验模式,包括在ATAC中通过测序进行选择抗原分析、基于噬菌体的单细胞多模态测序(PHAGE-ATAC)或DOGEMA-seq(一种同时捕获染色质可及性、细胞表面蛋白丰度和细胞谱系的信息的方法)。并且与单细胞RNA-seq相比较,mtscATAC-seq具有更好的潜在变异的整体覆盖率。虽然mtscATAC-seq具有诸多优势,但该方法是依赖于无法直接调节的自然mt DNA序列变异,同时对线粒体遗传动力学知之甚少,导致了在应用过程中可能存在一定的局限性。
总体来说,mtscATAC-seq为单细胞组学检测提供了一个更广泛的平台,弥补了体细胞突变检测的不足,该方法可用于线粒体遗传病以及致病性mtDNA的异质水平研究、利用线粒体DNA的变异进行谱系追踪以及造血系统动力学的评估。与传统的scATAC-seq相比,mtscATAC-seq具有相对均匀的线粒体基因组覆盖率,高测序深度以及富集度,且可同时与来自同一单细胞的细胞状态进行配对分析等优点。
[1] Stewart, J. B. & Chinnery, P. F. The dynamics of mitochondrial DNA heteroplasmy: implications for human health and disease. Nat. Rev. Genet. 16, 530–542 (2015).
[2] Kang, E. et al. Age-related accumulation of somatic mitochondrial DNA mutations in adult-derived human iPSCs. Cell Stem Cell 18, 625–636 (2016).
[3] Lareau, C. A. et al. Massively parallel single-cell mitochondrial DNA genotyping and chromatin profiling. Nat. Biotechnol. 39, 451–461 (2021).